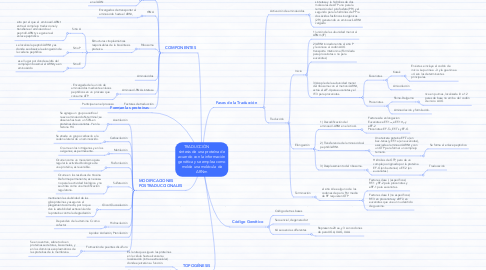
1. Forman las proteínas
2. COMPONENTES
2.1. mRNA
2.1.1. molécula de ácido nucleico de cadena sencilla que contiene la información genética almacenada en el ADN
2.2. tRNA
2.2.1. Encargados de transportar al aminoácido hasta el ARNr,
2.3. Ribosoma
2.3.1. Estructuras citoplasmáticas responsables de la biosíntesis proteica.
2.3.1.1. Sitio A
2.3.1.1.1. sitio por el que el aminoacil-ARNt entra al complejo traduccional y transfiere el aminoácido al peptidil-ARNt y se genera el enlace peptídico.
2.3.1.2. Sitio P
2.3.1.2.1. se localiza la peptidil-ARNt y es donde se observa la elongación de la cadena peptídica
2.3.1.3. Sitio E
2.3.1.3.1. es el lugar por donde saldrá del complejo ribosomal el ARNt ya sin aminoácido
2.4. Aminoácidos
2.5. Aminoacil-RNA sintetasa
2.5.1. . Encargada de la unión de aminoácidos mediante enlaces peptídicos en un proceso que consume ATP.
2.6. Factores de traducción
2.6.1. Participan en el proceso
3. TOPOGÉNESIS
3.1. Es la ruta que siguen las proteínas en la célula hasta alcanzar su localización (intra o extracelular) donde ejercerán su función.
3.2. El péptido señal y la región señal determinan el destino celu- lar correcto de las proteínas.
4. MODIFICACIONES POSTRADUCCIONALES
4.1. Acetilación
4.1.1. Se agrega un grupo acetilo al nuevo aminoácido N-terminal; se observa hasta en un 50% en proteínas de eucariotas. Y en la histona H4
4.2. Carboxilación
4.2.1. Se añade un grupo carboxilo a la cadena lateral de un aminoácido.
4.3. Metilación
4.3.1. Ocurre en los nitrógenos y en los oxígenos; es permanente.
4.4. Fosforilación
4.4.1. Ocurren como un mecanismo para regular la actividad biológica de una proteína, es reversible.
4.5. Sulfatación
4.5.1. Ocurre en los residuos de tirosina De forma permanente, es necesa- rio para la actividad biológica y no se utiliza como una modificación regulatoria.
4.6. Glico/Glucosilación
4.6.1. mantienen la solubilidad de las glicoproteínas y aseguran el plegamiento correcto, por lo que dan la estabilidad extracelular de la proteína contra la degradación.
4.7. Hidroxilación
4.7.1. Dependen de la vitamina C como cofactor
4.8. Lipidos: Acilación, Prenilación
4.9. Formación de puentes disulfuro
4.9.1. Se encuentran, sobre todo en proteínas secretoras, lisosomales, y en los dominios exoplasmáticos de las proteínas de la membrana.
5. Fases de la Traducción
5.1. Activación de aminoácidos
5.1.1. Mediante la aminoacil-ARNt sintetasa y la hidrólisis de dos moléculas de ATP uno para la remoción del pirofosfato (PPi) y el segundo para la hidrólisis de PPi a dos ácidos fosfóricos inorgánicos (2Pi) generando un aminoacil-ARNt cargado
5.2. Traducción
5.2.1. Inicio
5.2.1.1. 1) unión de la subunidad menor al ARNm (IF)
5.2.1.2. 2) ARNt iniciador entra al sitio P y reconoce al codón AUG transporta mtetionina (Formilada para procariotas o no para eucariotas)
5.2.1.3. 3) Acople de la subunidad menor del ribosoma con el metionil-ARNt, actúa el eIF-4 para eucariotas y el IF-3 para procariotas.
5.2.1.3.1. Eucariotas
5.2.1.3.2. Procariotas
5.2.2. Elongación
5.2.2.1. 1) Decodificación del aminoacil-ARNt en el sitio A.
5.2.2.1.1. Factores de enlongacion Eucariotas: eEF-1α, eEF-1βγ y eEF-2 Procariotas: EF-Tu, EF-Ts y EF-G
5.2.2.2. 2) Transferencia del aminoácido al peptidil-ARNt.
5.2.2.2.1. Una tercera poteina EF-Tu (en bac-terias) y EF-1a (en eucariotas), asociada al aminoacil-ARNt y con un GTP para formar un complejo ternario.
5.2.2.3. 3) Desplazamiento del ribosoma.
5.2.2.3.1. Hidrólisis de GTP, pero de un complejo originado por la proteína EF-G (en bacterias) o EF-2 (en eucariotas)
5.2.3. Terminación
5.2.3.1. el sitio A lee alguno de los codones de paro. Por medio de RF requieren GTP
5.2.3.1.1. Factores clase I, (específicos) RF-1 y RF-2 para procariotas y eRF-1 para eucariotas.
5.2.3.1.2. Factores clase II (no específicos) RF-3 en procariotas y eRF-3 en eucariotas que une un nucleótido de guanina.
6. Código Genético
6.1. Código de tres bases
6.2. Secuencial, degenerado.t
6.3. 64 secuencias diferentes
6.3.1. Representa 20 aa, y 3 son codones de paro UGA, UAG, UAA.
